Tartalomjegyzék:
- 1. lépés: Agyi doboz használata
- 2. lépés: Bevezetés: Keresztmetszetek elemzése
- 3. lépés: A funkció beállítása: LevelCurveTracings.m
- 4. lépés: A kötött algoritmus kifejlesztése: határok keresése
- 5. lépés: A kötött algoritmus fejlesztése: külső kötött tömb létrehozása
- 6. lépés: A kötött algoritmus kidolgozása: Munka a középponttal
- 7. lépés: A kötött algoritmus kifejlesztése: Ha egy centroid nincs középen
- 8. lépés: A kötött algoritmus fejlesztése: lyukak interferenciája
- 9. lépés: A kötött algoritmus kidolgozása: lyukak keresése, agyi határok és lyukak véglegesítése
- 10. lépés: Adatok naplózása: Funkció PatientFiles.m
- 11. lépés: Adatok naplózása fájlokba
- 12. lépés: Adatok naplózása: Az agy térfogatábrájának megjelenítése az idő múlásával
- 13. lépés: A részterületek hiányosságainak megszüntetése: Subplotclose.m
- 14. lépés: A fő kód: Az összes törlése és a bemenetek kérése
- 15. lépés: A fő kód: A képek kötegelt feldolgozása
- 16. lépés: A fő kód: párnázás
- 17. lépés: A fő kód: határok meghatározása
- 18. lépés: A fő kód: A megfelelő Z méret meghatározása
- 19. lépés: A fő kód: X és Y koordináták meghatározása
- 20. lépés: A fő kód: Háromdimenziós struktúra ábrázolása, kötet keresése és adatok naplózása
- Szerző John Day day@howwhatproduce.com.
- Public 2024-01-31 10:20.
- Utoljára módosítva 2025-01-23 14:48.
A hosszabb emberi élet határvidékére való előretörés előidézte a civilizációk által nem látott betegségek kialakulását a miénk előtt. Ezek közül az Alzheimer -kór megközelítőleg 5,3 millió élő idős amerikait érintett 2017 -ben, vagy körülbelül minden tizedik idős amerikait (https://www.alz.org/facts/) és számtalan más demenciát. Ez a kód segíti a harcot, hogy megértsük, mi sújtja vénjeinket, ez a kód a jövő kutatóit és kíváncsi kíváncsiságait képes lesz az agy térfogatának időbeli követésére.
1. lépés: Agyi doboz használata
Az agydoboz használatához csak a következőkre van szükség:
- Az agy MRI -vizsgálata és az ilyen fájlok neve és formátuma (nagyjából azonos méretekkel kell rendelkezniük)
- Egy szkennelés hossza
- Az egyes rétegek közötti távolság (MRI -vizsgálat)
- Páciens neve (bevitelkor ne tegyen szóközt, és kérjük, írja nagybetűvel a kereszt- és vezetéknevet, például: Keresztnév Utónév)
Ebből pedig az ember képes nyomon követni az egyének agyi térfogatának időbeli alakulását. Így ez a szoftver nyomon tudja követni az Alzheimer -kór tendenciáit. A vizsgálat során használt hossza 180 mm volt egy vizsgálat hossza és 5 mm az MRI vizsgálatok közötti távolság, az átlagos adatok alapján.
Az agydoboz alkalmazását azonban nem kell erre az egyetlen feladatra korlátozni. Ha egy adott szilárd anyag keresztmetszete fénykép, mint egy daganat önmagában, akkor ezek térfogatváltozásának tendenciái is nyomon követhetők a szoftverben.
2. lépés: Bevezetés: Keresztmetszetek elemzése
Háromdimenziós szerkezetekben a kétdimenziós síkokat, amelyekből állnak, keresztmetszeteknek nevezzük. Képzelje el, hogy egy halom papír egy téglalap alakú prizmát alkot, akkor minden papír a papír keresztmetszete lesz. Az agy elképzelésében ugyanazt a gondolatmenetet alkalmazzuk. Az MRI (mágneses rezonancia képalkotás) (lásd az MRI -vel kapcsolatos információkat) rögzíti az agy keresztmetszetét, és az agy minden egyes "rétegében" meghatározott határok felhasználásával felépíthetünk egy szerkezetet az agy térfogatának modellezésére és megtalálására. Először azonban létre kell hoznunk egy funkciót, amely információt nyújt az ilyen határokról.
3. lépés: A funkció beállítása: LevelCurveTracings.m
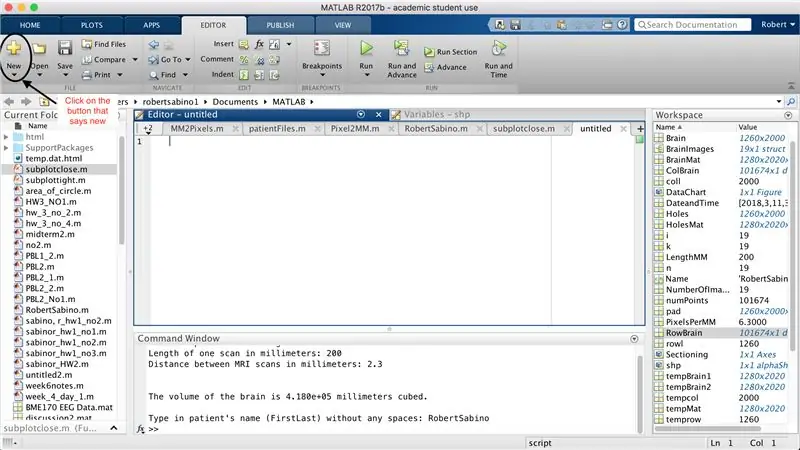
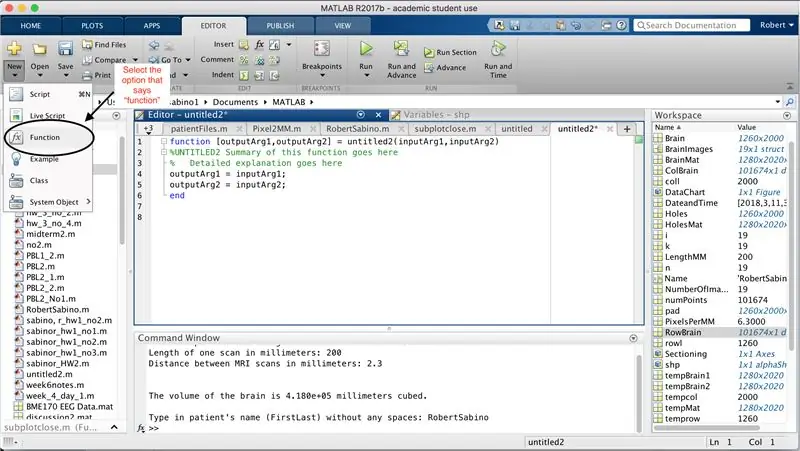
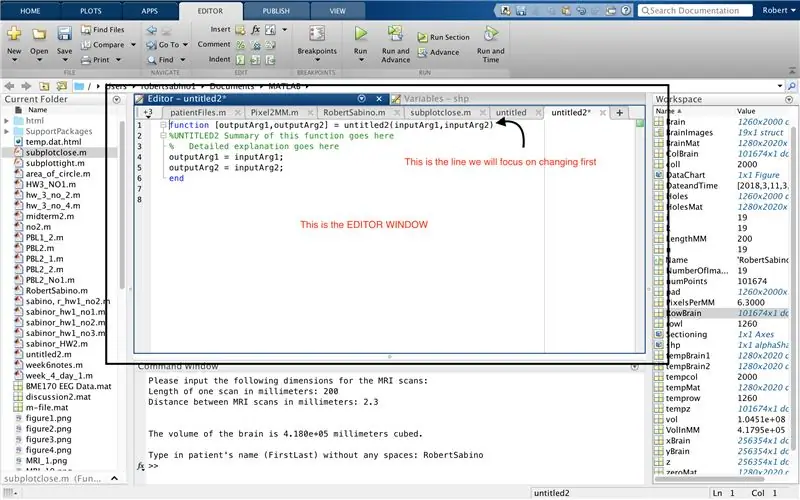

Először győződjön meg arról, hogy a számítógépére letöltött MATLAB_R2017b letölthető (töltse le itt), és nyissa meg a MATLAB alkalmazást. A MATLAB kezelőfelületén kattintson az ablak bal felső sarkában található gombra, amelyen az "Új" felirat látható, vastag sárga pluszjelekkel, majd válassza a "funkció" lehetőséget, hogy a szerkesztőablakban a térhez hasonló tér nyíljon meg. harmadik kép. A funkció beállításához az első sor megváltoztatására összpontosítunk. Ahol azt írja, hogy "outputArg1", cserélje ki "agy" -ra, "outputArg2" -re, hogy "hole" -ot, "untitled2" -et "exp2" -re és "inputArg1" -et "image" -re mondjon, és törölje az "inputArg2" parancsot. Most egy függvényt kell meghívni az "exp2" használatával, egy argumentumot "image" kell venni, és ki kell írni az "agy" és a "lyukak" határait. A függvény első sora hasonlít a negyedik képen látható vonalra. Törölje az összes kódot a kezdő sor alatt.
4. lépés: A kötött algoritmus kifejlesztése: határok keresése

Írja be a kódot a sor alatt. A függvény ezen szakasza soronként a következőt hajtja végre.
- Töltse be az "image" képet az "mri" változóba.
- Változtassa az "mri" -et olyan képpé, amely a számok egytől számig terjedő értékeiből áll össze (nulla) (más néven bináris) a beállított küszöbérték alapján. Ha egy képpont értéke 0,1 vagy annál nagyobb, akkor egy, ha nem, akkor az adott pixel értéke nulla.
- A következő négy sor 10 oszlopot és sort az MRI -szkennelési réteg szélein nullává változtat, hogy elkerülje a helytelen értékek határértékként való olvasását (amint azt a kóddal végzett kísérletekből megtudtuk).
- Az utolsó sorban a bwboundaries nyomon követi az "mri" bináris kép határait, és egyenlővé teszi "b" -vel, egy tömböt, amelynek elemei megegyeznek az indexekkel.
5. lépés: A kötött algoritmus fejlesztése: külső kötött tömb létrehozása
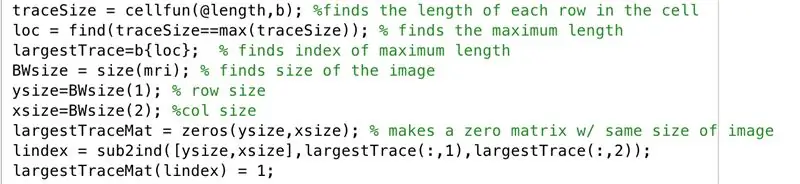
Kövesse a szerkesztőablakban a következő kódot a képen. A kód ezen szakasza soronként a következőket teszi.
- Keresse meg a "b" bináris kép minden sorának hosszát (a cellfun minden sorra alkalmazza a függvény hosszát).
- Állítsa a "loc" beállítást a maximális hosszúságok tárolására.
- Keresse meg a maximális hossz indexét, amely a "legnagyobbTrace" -ben tárolásra van beállítva.
- Keresse meg a "mri" kép méretét, amely ugyanabból a méretből áll, mint a "b", és állítsa a "BWsize" értékre.
- Keresse meg a sorok számát a kép tömbjében, állítsa "ysize" értékre.
- Keresse meg az oszlopok számát a kép tömbjében, állítsa "xsize" értékre.
- Létrehoz egy "legnagyobbTraceMat" tömböt, egy "ysize" az "xsize" mátrix nullákat.
- Keresse meg az egyenértékű indexet az előjegyzett értékek közül, ahol a legnagyobbTrace x értékek és y értékek voltak, és tárolja a "lindex" vektorban.
- A nullák mátrixában, a "legnagyobbTraceMat" -ban, a "lindex" -ben elemként tárolt indexértékekhez tartozó indexek elemeit alakítsa eggyé.
Így a "legnagyobbTraceMat" logikai tömb az adott agyszkennelés keresztmetszetének legnagyobb határolt régiójával van ábrázolva, nullák hátterével
6. lépés: A kötött algoritmus kidolgozása: Munka a középponttal

Ezután meg kell vizsgálnunk, hogy a keresztmetszet több régióból áll -e (a legnagyobb). A legnagyobb régió centroidja igazításának tesztelésével láthatjuk, hogy van -e egy szomszédos régió, amely jobban centrált centroidot eredményezne, vagy több régió lehetősége.
- A "regionProps" használatával keresse meg az információkat a jelenlévő középpontokról, a "tempStruct" struktúrával egyenlőnek beállítva
- Formálja a "centroidok" tömböt a "centroid" mező adataival függőlegesen összefűzve
- Vegyük a "középpontok" második oszlopértékeit (a vízszintes méretkoordinátákat)
- Futtasson egy szűrőt, hogy ellenőrizze a centroid vízszintes középponthoz igazítását
7. lépés: A kötött algoritmus kifejlesztése: Ha egy centroid nincs középen
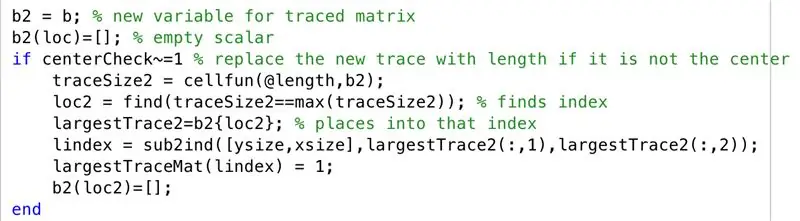
Abban a forgatókönyvben, hogy a legnagyobb nyomkövetési régió középpontja nincs középen, a következő lépéseket hajtjuk végre. Amint azt az MRI -felvételek során megfigyeltük, az a tendencia, hogy az agyféltekéket a keresztmetszetben ábrázolják, ha nem szomszédosak, ezért most folytatjuk a második legnagyobb nyomvonal ábrázolását a "legnagyobbTraceMat" legnagyobb nyomával együtt
- Állítsa be a nyomkövetett mátrixot egy új "b2" változóra
- Inicializálja az üres "b2" mátrixot, a "loc" indexelésű halmazzal
- Hozzon létre egy feltételt, ha egy centroid nincs középen (azaz többrégiós réteg)
- Állítson be minden nyomon új nyomkövetési méretet (traceSize2)
- A "loc2" beállításával keresse meg azokat az indexeket, ahol a határok jelen vannak
- Legyenek a "loc2" által a "b2" -ben megadott cellák a "legnagyobbTrace2" -vel
- Alakítsa át az indexeket indexekbe, állítsa "lindex" -re
- Módosítsa a "legnagyobbTraceMat" "lindex" -nek megfelelő elemeket 1 -re
- Inicializálja az üres "b2" mátrixot, a "loc2" indexelt halmazzal
8. lépés: A kötött algoritmus fejlesztése: lyukak interferenciája

A lyukak kezelésekor a "b2" -ben tárolt értékek nyomon követik a legnagyobb nyomon kívüli struktúrákat, és ezeket a "legnagyobbTraceMat" kitöltött formájára ábrázolva kiderül, hol vannak lyukak az agyterületeken.
- Hozzon létre tömböt "fillMat", amely a "legnagyobbTraceMat" kitöltött formája
- Hozzon létre "interferenceMat" tömböt, "ysize" x "xsize" tömböt
- Hozzon létre "interferenceloc" tömböt a "b2" értékek függőleges összefűzéséhez
- Hozzon létre "lindex" tömböt az "interferenceloc" -nak megfelelő indexek tárolására
- Az "interferenciaMat" indexek esetén, amelyek "lindex" -nek felelnek meg, állítsa az értéket 1 -re, és készítsen egy másik határolt régiót
9. lépés: A kötött algoritmus kidolgozása: lyukak keresése, agyi határok és lyukak véglegesítése

- Állítsa a "tempMat" tömböt az "interferenceMat" plusz "fillMat" értékre, így a mátrix minden értékét egymáshoz adja hozzá
- Állítsa be a "holeLoc" tömböt azokkal az indexekkel, ahol az "interferenceMat" és a "illedMat "egyaránt egyenlő
- A "holesMat" beállítása nulla mátrixként "ysize" x "xsize"
- Állítson be indexeket a "holeMat" -ba, amelyek egyenlők a "holeLoc" értékkel
- Az "agy" beállítása "legnagyobbTraceMat" értékre
- Állítsa a "lyukak" értéket "holeMat" értékre
Azzal a megállapítással, hogy a hozzáadott mátrixok értéke 2 volt, a lyukak helyét könnyen rögzítették és egy üres mátrixra rajzolták.
10. lépés: Adatok naplózása: Funkció PatientFiles.m
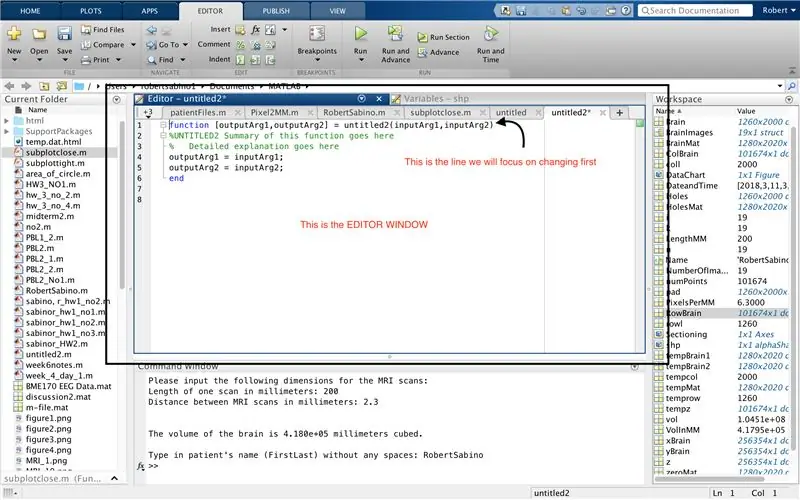

Az utolsó funkció beállításához hasonlóan kattintson az ablak bal felső sarkában lévő gombra, amelyen az "Új" felirat látható, vastag sárga pluszjelekkel, majd válassza a "funkció" lehetőséget, hogy a szerkesztőablakban megnyisson egy helyet, amely hasonlít a harmadik képre. Az első sorban törölje a kimeneti mátrixot, és csak „output” -ra cserélje ki, az „untitled2” -et cserélje ki a „patientFiles” -re, törölje az összes bemeneti argumentumot, és kövesse a kódsor negyedik képén megadott formázást. Ennek a funkciónak az első sorának meg kell egyeznie a kép formázásával.
11. lépés: Adatok naplózása fájlokba
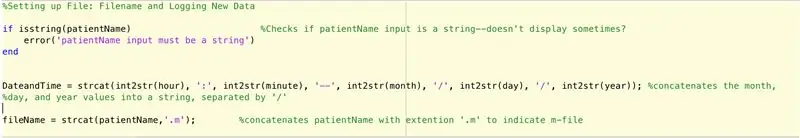

Ahhoz, hogy beállíthassunk egy fájlt a főfunkció által talált (még le nem írt) adatok naplózására, ezeket a lépéseket kell követnünk (a kód soronként előírtak szerint).
- Ellenőrizze, hogy a betegnév bemenete karakterlánc.
- Ha nem karakterlánc, jelenítse meg, hogy a betegnév bemenet karakterlánc.
- Fejezze be az if utasítást (megelőzze a hibát).
- Állítson be egy "DateandTime" karakterlánc-utasítást, amely a következő formátumot adja meg: óra: perc-hónap/nap/év.
- Állítsa be a fileName változót a következőre: betegName.m.
Most a függvény következő szakaszához: Létezik már ilyen nevű fájl?
1) Tegyük fel, hogy az ilyen nevű fájl már létezik:
- Futtassa a fájlt a múltbeli értékek lekéréséhez
- Adja hozzá az aktuális iteráció "DateandTime" adatait új cellaként az x értékű cellatömbhöz (index vége+1)
- Adja hozzá az aktuális "brainVolume" értéket új cellaként az y értékű cellatömbhöz (index vége+1)
- Mentse a fájlba betöltött aktuális változókat.
2) Tegyük fel, hogy az ilyen nevű fájl nem létezik:
- Hozzon létre egy új fájlt a "betegnév" változóban tárolt névvel
- Adja hozzá az aktuális "DateandTime" adatokat cellaként az x érték üres cellatömbéhez
- Adja hozzá az aktuális "brainVolume" adatokat cellaként az y érték üres cellatömbéhez
- Mentse a fájlba betöltött aktuális változókat.
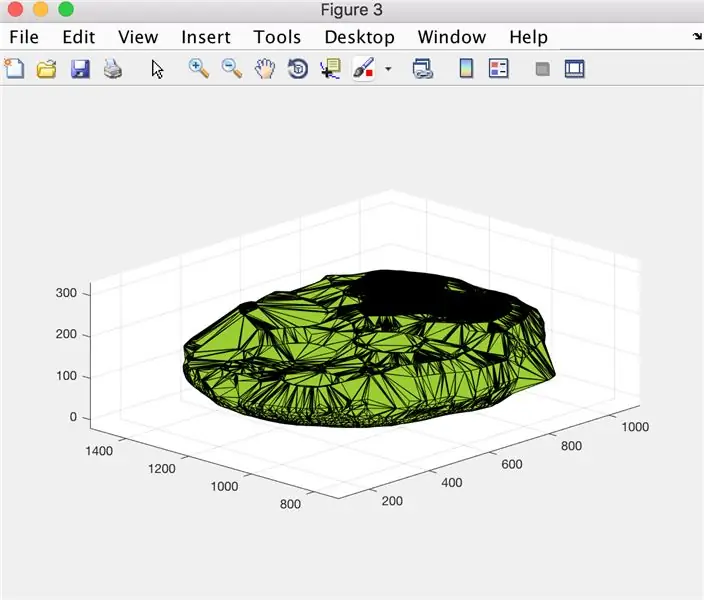
12. lépés: Adatok naplózása: Az agy térfogatábrájának megjelenítése az idő múlásával

- Konvertálja az x értékek tömbjét (xVals) kategorikus tömbvé (xValsCategorical), hogy lehetővé váljon a rajzolás
- Ábraablak létrehozása 5
- Ábrázolja az "xValsCategorical" és az "yVals" által kijelölt pontokat (amelyek agytérfogatot tartalmaznak), üreges körökkel jelezve a pontokat és szaggatott vonallal összekötve
- A cselekmény címe: betegName Agymennyiség adatai
- Címkézze fel az x tengelyt a képen látható módon
- Címkézze fel az y tengelyt a képen látható módon
- Az 5. ábra legyen egyenlő a kimenettel
Ebből a betegnév függvény meghívása egy fájlt eredményez, amelynek szerkesztett adatai nyomon követik az agy térfogatát az idő múlásával, és ábrázolja a trendeket.
13. lépés: A részterületek hiányosságainak megszüntetése: Subplotclose.m

A https://www.briandalessandro.com webhelyről származó kódból adaptált függvény az MRI képeket és az agyrétegeket létrehozó számok létrehozásakor arra szolgál, hogy lezárja a réseket a fő kód alrészei között. A subplotclose.m -en belül használt részterület -funkció az adott részterületek helyzetét úgy állítja be, hogy a hosszabb dimenzió szempontjából szorosan illeszkedjenek egymáshoz. Például, ha a kód 7 x 3 mátrixot szán, a sorok illeszkednek egymáshoz, mivel a sor mérete hosszabb. Ha a kód 3 x 7 mátrixot szándékozik adni, az oszlopok szorosan illeszkednek, a sorokban rések vannak, ahogy az a fő kódunk ábráin látható.
14. lépés: A fő kód: Az összes törlése és a bemenetek kérése

A fő kód elindításához kattintson ugyanazon a gombra, amely az ablak bal felső sarkában az "Új" feliratot írja, és válassza a "Szkript" lehetőséget a "Funkció" helyett a korábbi szakaszokból. Írja be a kódot a képen látható módon a szerkesztőablakban. A kódsorok a következő feladatokat látják el sorrendben:
- Zárja be az összes megnyitott fájlt a 0, 1 és 2 kivételével.
- Zárjon be minden ábraablakot.
- Törölje az összes változót a munkaterületen.
- Törölje a parancsablakot.
- Megjelenítés a parancsablakban: Kérjük, adja meg a következő méreteket az MRI -vizsgálatokhoz:
- A parancsablak új sorában kérdezze meg: Egy letapogatás hossza milliméterben:. A felhasználó által megadott válasz a "lengthMM" változó lesz.
- Új sorban kérdezze meg: Az MRI -vizsgálatok közötti távolság milliméterben:. A felhasználó által megadott válasz a "ZStacks" változó lesz.
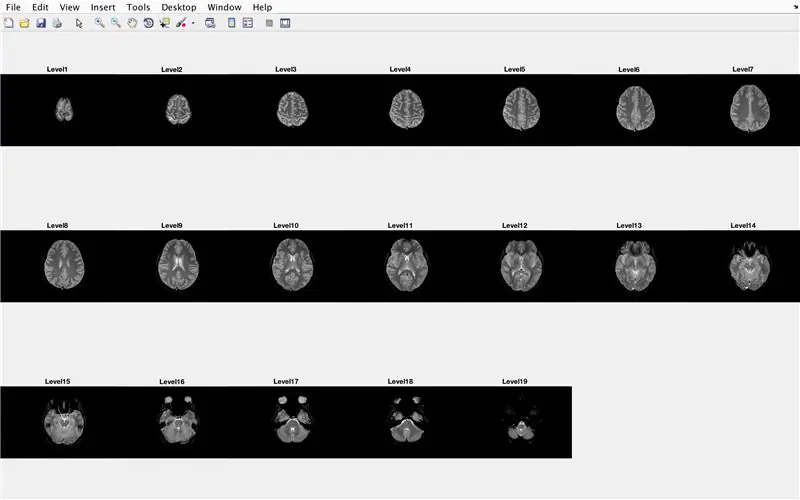
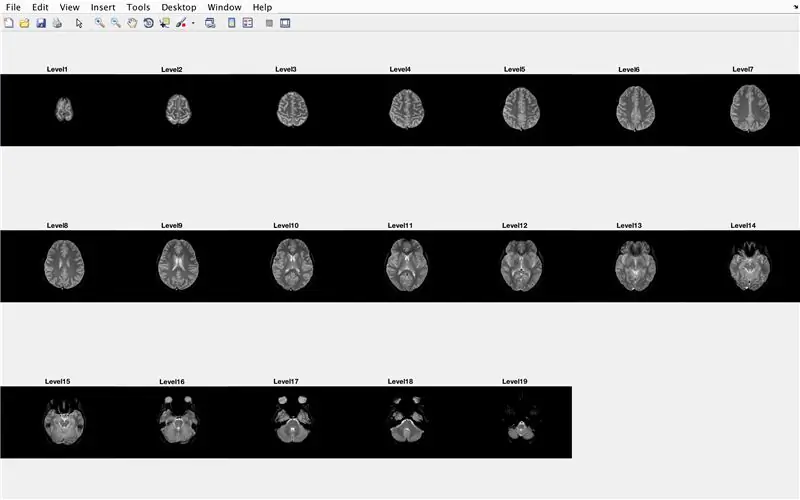
15. lépés: A fő kód: A képek kötegelt feldolgozása

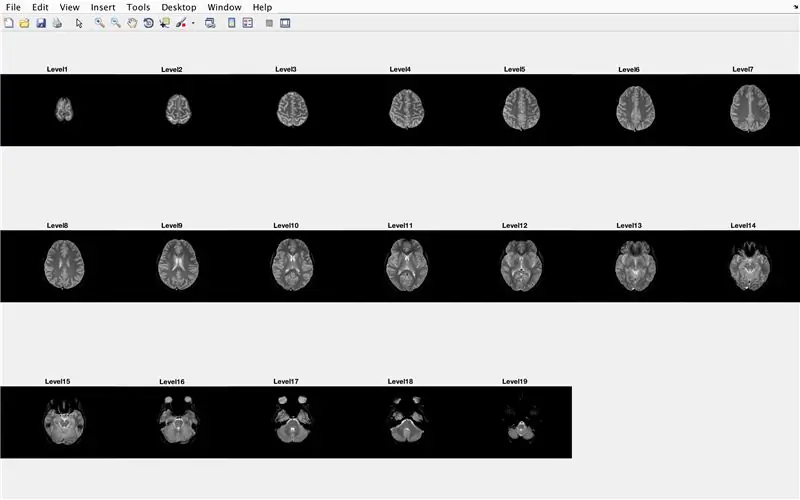
Ebben a részben a kód betölti a képeket (amelyek az agy keresztmetszeteinek MRI -vizsgálatából állnak), és minden képfájl nevét a "Base" változóban tárolja, és megjeleníti az MRI -vizsgálatokat. Kérjük, kövesse a képen látható kódot, amely a következőket teszi:
- Hozzon létre "BrainImages" struktúrát, amely információkat tartalmaz az aktuális mappában található összes fájlról, amelyek illeszkednek az MRI névformátumához _ (). Png
- Állítsa be a "NumberofImages" változót a "BrainImages" szerkezet tömb elemeinek számával
- Nyissa meg az ábra ablakot 1
- Állítsa be a ciklus ciklusát a fájlban megszámlált képek számához
- Minden ciklus esetében a "CurrentImage" az egyes fájlok megfelelő neve MRI_i.png, az iterációs szám "i"
- Hozzon létre egy 3x7 -es részterületet az "imshow" által betöltendő 19 kép megjelenítéséhez
- Jelenítse meg az egyes képeket egy másik elemként az aldiagram ablakban
- Nevezzen minden alrészletet Level_ -nek, ahol üres a for ciklus iterációs száma.
- A for ciklus befejezése (a hiba elkerülése érdekében)
Ez megjeleníti az 1. ábraablakban az összes MRI vizsgálatot nyers formában, 3 x 7 konfigurációban, az x tájolás hiányában.
16. lépés: A fő kód: párnázás

A párnázással elkerüljük a képméretek enyhe eltéréseit, amelyek hibát okozhatnak a méretbeli eltérések esetén, ha az egyik kép valamivel nagyobb, mint a másik.
- Nyissa meg az ábra ablakot 2
- Töltse be a képmátrixot az MRI_1-p.webp" />
- Keresse meg a kép mátrixának méretét, és állítsa az "OriginalXPixels" (a sorok száma) és az "OriginalYPixels" (az oszlopok száma) értékre
- Állítsa be a "BrainMat" mátrixot úgy, hogy minden nullából álljon, további 20 sorból és további 20 oszlopból minden síkban, valamint 19 teljes keresztmetszetből, egy -egy síkonként.
- Állítsa be a "HolesMat" -t, hogy ugyanabból a háromdimenziós nullákból álljon, és később adja meg a lyukkoordinátákat
- Hozzon létre egy "zeroMat" -t, aminek mérete a pad, plusz húsz sor és húsz oszlop, egy kétdimenziós nullák tömbje.
17. lépés: A fő kód: határok meghatározása
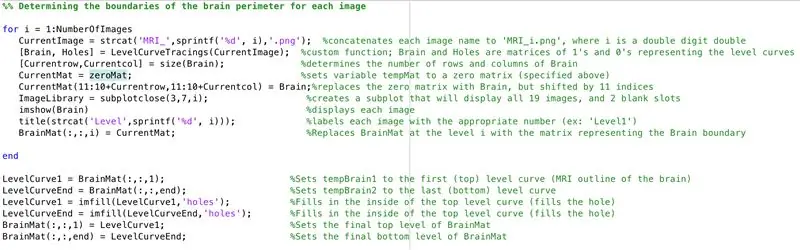
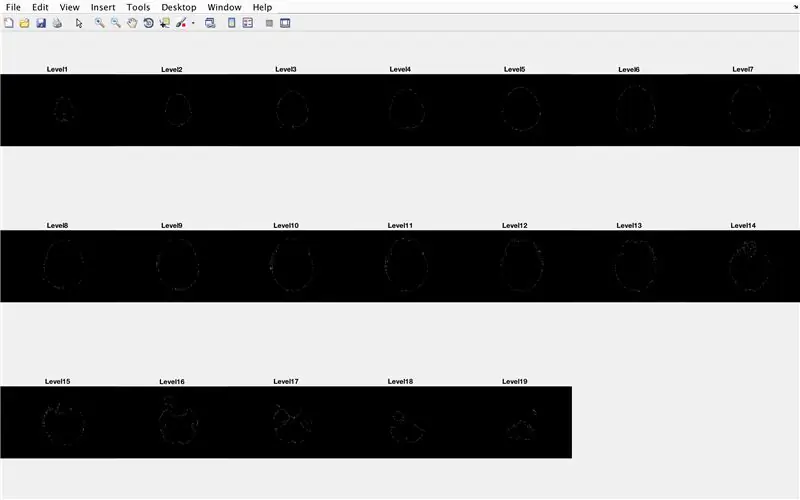
- Állítsa be a for ciklust, hogy végigmenjen a korábban betöltött képek adatain
- Hasonló módon, a kötegelt feldolgozás során a "CurrentImage" betölti az "MRI_i.png" fájlokat, ahol i az iterációs szám
- Futtasson minden képet a korábban létrehozott "LevelCurveTracings2.m" feldolgozási funkción keresztül
- Keresse meg a "Brain" kimenet méretét, állítsa a sorok számát "Currentrow" értékre, az oszlopok számát pedig "Currentcolumns" értékre
- Állítsa a "CurrentMat" nullák mátrixára a "Currentrow" és az "Currentcolumns" által megadott méretekkel
- Központosítsa az "Agy" adatait a "CurrentMat" -be, 10 soros margóval minden oldalon
- Hozzon létre egy 3 x 7 -es méretarányt a képek határainak megjelenítéséhez
- Adjon címet az ábra ablakának minden részrészletéhez
- Hozzon létre háromdimenziós „BrainMat” mátrixot, amely minden „CurrentMat” határrétegből áll
- A for ciklus befejezése (a hibák elkerülése érdekében)
A következő alfejezet kitölti a javasolt háromdimenziós forma tetején és alján maradt lyukakat
- Állítsa be a "LevelCurve1" értéket a "BrainMat" első rétegével (szilárd anyag alja)
- Állítsa be a "LevelCurveEnd" értéket a "BrainMat" utolsó rétegével (szilárd anyag teteje)
- Írja felül a "LevelCurve1" -t egy kitöltött réteggel
- Írja felül a "LevelCurveEnd" kitöltött réteggel
- Állítsa a kitöltött réteget a "BrainMat" alsó rétegére
- Állítsa a kitöltött réteget a "BrainMat" felső rétegére
18. lépés: A fő kód: A megfelelő Z méret meghatározása

Az első három sor egy üres "z" tömb beállításából és egyszerű konverziós műveletekből (a képpontok hosszának elosztásából) áll, hogy a térfogatot megfelelően mm -ben leolvassuk^^3
- Hozzon létre egy for ciklust, hogy végigjusson az egyes rétegeken
- Keresse meg az egyes rétegek számát egy adott rétegben
- A z koordinátákat alakítsa át a megfelelő arányra méretezett értékekké, "tempz" értékre, oszlopvektorra
- Adjon hozzá z értéket a szintgörbéhez a z vektorhoz
Ezzel a z koordinátákat megfelelően állítja be.
19. lépés: A fő kód: X és Y koordináták meghatározása

Most határozza meg a határok egyes pontjainak x és y pozícióját.
- Inicializálja az "xBrain" -t üres tömbként
- Inicializálja az "yBrain" -t üres tömbként
- Állítson be egy hurkot a betöltött képeken való ciklushoz
- Fordítson össze két oszlopos mátrixot, hogy tárolja az egyes pontok síkbeli koordinátáit a korlátban, amelyeket az "RowBrain" és az "ColumnBrain" oszlopvektorok képviselnek.
- Az "xBrain" csatolása az aktuálisan talált "RowBrain" koordinátákkal
- Az "yBrain" hozzáfűzése a jelenleg talált "ColumnBrain" koordinátákkal
- A for ciklus befejezése (a hiba elkerülése érdekében)
20. lépés: A fő kód: Háromdimenziós struktúra ábrázolása, kötet keresése és adatok naplózása
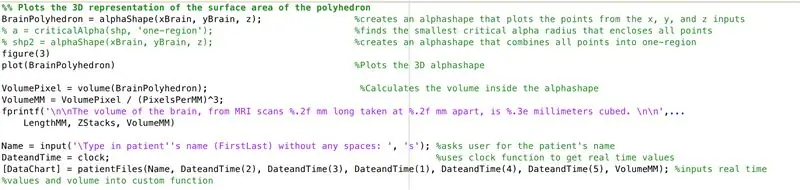

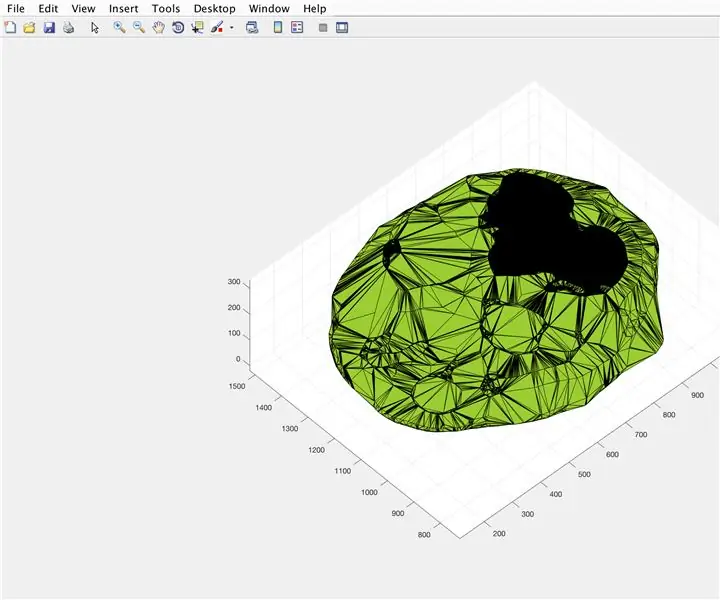
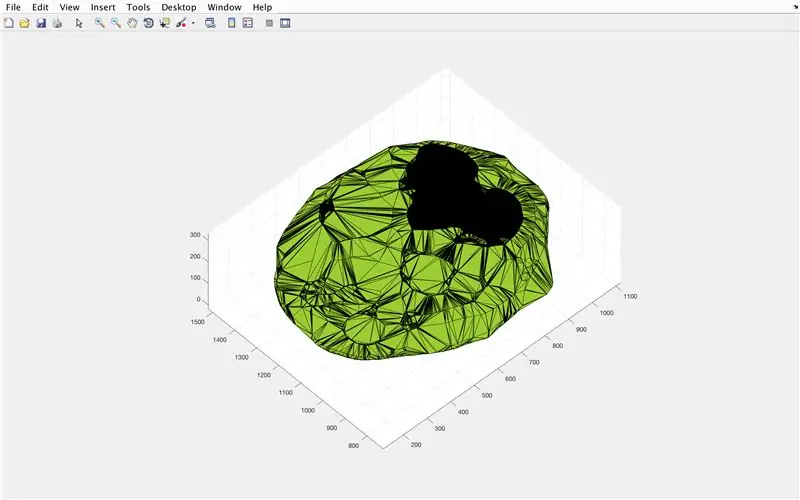
Az alphaShape függvény használatával létrehozunk egy háromdimenziós szerkezetet, amelyből kiszámíthatjuk az agy térfogatát.
- Használja az alphaShape függvényt, csatlakoztassa az "xBrain", "yBrain" és "z" vektorokat az x, y és z koordinátákhoz, és állítsa be a "BrainPolyhedron" értéket.
- Nyissa meg az ábra ablakot 3
- Ábrázolja a kiszámított "BrainPolyhedron" alfa alakzatot, és jelenítse meg az ábra ablakban
- Számítsa ki az alfa alakzat térfogatát az alfa alakzatokhoz használható "volume" függvény használatával
- Alakítsa át a hangerőt mm^3 -ra
- Nyomtassa ki a szilárd anyag térfogatát a Parancsablakban
- Kérje meg, hogy a beteg nevét állítsa be bemenetként
- Szerezze be az aktuális dátumot és időt az órával, és állítsa a "DateandTime" értékre
- Hívja fel a "patientFiles" funkciót a számított adatok naplózásához és ábrázolásához
Innen a második és a harmadik képen a felbukkanó ábrákat, a negyediken pedig a parancsablakban megjelenítendő ábrákat kell megjeleníteni.
Ajánlott:
Arduino GPS óra helyi idővel a NEO-6M modul használatával: 9 lépés
Arduino GPS óra helyi idővel a NEO-6M modul használatával: Ebben az oktatóanyagban megtanuljuk, hogyan lehet pontos időt lekérni a műholdakról az arduino használatával. Nézze meg a videót
"A világ legegyszerűbb" neurális szerkezete (Men in Black Memory Eraser): 10 lépés (képekkel)

"Világ legegyszerűbb" Neuralizer-build (Men in Black Memory Eraser): Jársz jelmezpartira néhány nap múlva, de még mindig nincs jelmezed? Akkor ez a konstrukció az Ön számára! Napszemüveggel és fekete öltönyvel ez a kellék teszi teljessé Férfi fekete ruháját. A lehető legegyszerűbb elektronikus áramkörön alapul
Játék az idővel: 6 lépés

Játék az idővel: A projekt kezdeti ötlete a következő volt: készítsen egy ajándékot, hozzon létre egy eredeti elektronikus áramkört, és használjon egy régi telefonhívást, és " steampunk " megjelenés
Ez egy kéz? (Raspberry Pi kamera + neurális hálózat) 1/2 rész: 16 lépés (képekkel)

Ez egy kéz? (Raspberry Pi Camera + Neural Network) 1/2 rész: Néhány nappal ezelőtt az edzőteremben megsérült a jobb csuklóm. Később minden alkalommal, amikor a számítógépes egeret használtam, sok fájdalmat okozott a meredek csuklószög miatt. Ekkor ütött belém " nem lenne jó, ha bármilyen felületet átalakíthatnánk nyomkövetővé
Arduino neurális hálózati robot: 21 lépés (képekkel)

Arduino neurális hálózati robot: Ez az oktatható anyag a Make YouTube Channel számára készített 3 részből álló sorozaton alapul, amely pontosan bemutatja, hogyan kell prototípusozni, tervezni, összeszerelni és programozni saját Arduino neurális hálózati robotját. A teljes sorozat megtekintése után jobbnak kell lennie
