Tartalomjegyzék:
- Szerző John Day day@howwhatproduce.com.
- Public 2024-01-30 09:40.
- Utoljára módosítva 2025-01-23 14:47.
Sandbox projektünk célja, hogy segítse a biológiai területen dolgozó kutatókat a sejtminták elemzésében és sejtjeik állapotának kiderítésében. Miután a felhasználó beírt egy képet a cellamintájáról, kódunk feldolgozza a képet, hogy készen álljon a cellaszámlálásra a kép szürkeárnyalatosra és binárisra konvertálásával. A kód küszöbértéket használ a tényleges cellákra vonatkozó, többletinformációk eltávolítására, hogy pontosan megtalálja a sejt tömegeit. A képen lévő cellák számának megszámlálása után a kódunk megadja a kép pixelméretét, majd megtalálja a cellaterület eloszlását a cellák számának és a sejtek területének függvényében történő ábrázolásával annak érdekében, hogy a felhasználó az összefolyást adja. a tenyésztő edény felületének százalékos aránya, amelyet az érdeklődő sejtek borítanak. Az összefolyás alapján a kutató megállapíthatja, hogy a sejteket passzálni kell -e vagy sem; a sejtek áthaladása a sejtek vagy mikroorganizmusok életének vagy számának kiterjesztésére utal, ha a tenyészet egy részét friss növekedési táptalajra visszük át, és ez hasznos, ha a sejtek túl nagyra nőnek vagy elfogynak az élelmiszerek. A kód futtatása és a kép feldolgozása után a felhasználó eldöntheti, hogy elfogadja vagy elutasítja az eredményeket, és szükség esetén módosíthatja a küszöböt az adatok jobb lekérése érdekében.
1. lépés: Válassza ki és konfigurálja a képet az elemzéshez
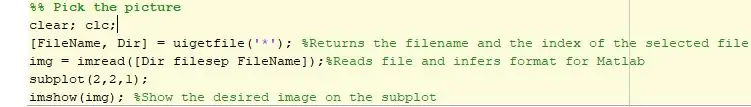
A kódunk első lépése a megfelelő kép kiválasztása és konfigurálása a Matlab elemzéséhez. Kiválaszthatjuk a képet az uigetfile funkció segítségével, amely lehetővé teszi, hogy bármilyen fájlt megragadjunk és beilleszthessünk a programba. Ezután az imread használatával olvassuk el a képet, és konfiguráljuk a Matlab elemzéséhez. A kiválasztott kép ezután megjelenik egy részterületen.
2. lépés: Küszöbérték és GUI
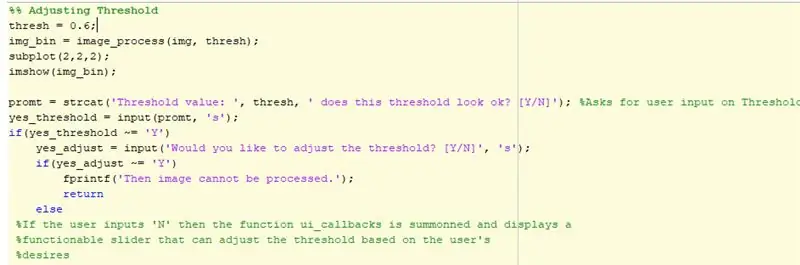
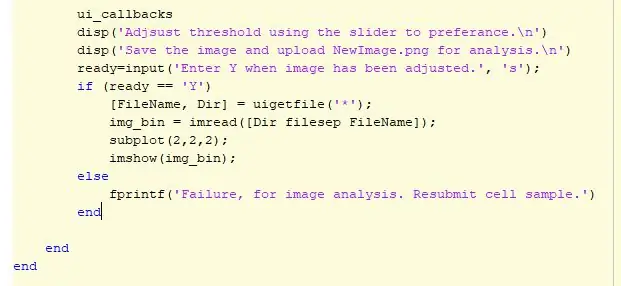
A kód legelején a képet az „uigetfile” segítségével választjuk ki, majd ezt a képet egy változóval definiáljuk. A változó ezután a kód azonosítására szolgál különböző elemzések futtatásakor. Az ábrán 2x2 alterület jön létre. Az 1. pozícióban az eredeti kép jelenik meg. A kód következő része a küszöbértékek kiigazításának helye. Kezdetben egy 0.6 -os küszöbértéket használnak, és az alterület 2. pozíciójában jelenik meg. Az if-utasítás ezután határozza meg, hogy a felhasználó meg akarja-e tartani a küszöböt, vagy módosítani kívánja. A felhasználó beállíthatja a küszöböt egy olyan grafikus felhasználói felület segítségével, amely tartalmazza a különböző küszöbértékű képeket, a csúszkát és a mentés gombot. A küszöbérték beállítása után a felhasználó a mentés gombra kattintva elmenti a képet, és azt a felhasználók MATLAB fájljaiban tárolja-p.webp
3. lépés: Kontúrok ábrázolása és sejteloszlás
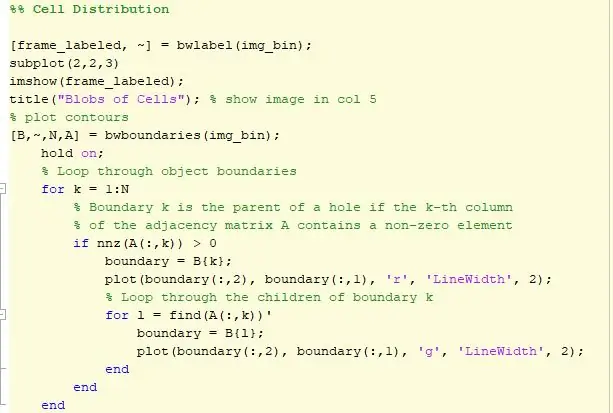
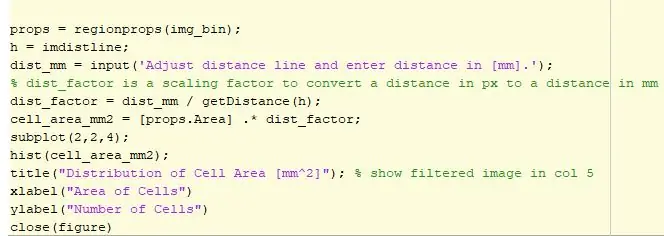
A kód következő része a kontúrokat ábrázolja. Van egy hurok, amelyben a cellákat vörös kerület veszi körül, és azok a cellák, amelyek egy másik cella tetején vannak, zölddel vannak körvonalazva. A körvonalazott kép ezután interaktív távolságvonallal jelenik meg a 3. pozícióban. Ez a sor határozza meg a vonalban lévő képpontok számát, amelyet a felhasználó beállított egy képpont -milliméter átalakítóhoz. A távolságtényezőt ezután megszorozzuk a területprofilokkal meghatározott területtel, és a területet milliméter négyzetben fejezzük ki. Az adatokat ezután hisztogram segítségével ábrázoljuk, hogy lássuk a sejtek területi eloszlását. Ez a hisztogram ezután megjelenik a 4 -es pozícióban.
4. lépés: A cellakép konvertálása

Ebben a lépésben felvettük a szürkeárnyalatos képet, és binarizáltunk, szűrtünk és fordítottunk. Ha ezeket a funkciókat a képen elvégezte, eltávolította a zajos pixeleket, amelyek összetéveszthetők a cellákkal, és simábbá és puhábbá tette a képet a cella élei körül. Ezt azért tették, hogy a képen lévő sejteket megkülönböztethető "foltként" nyerjék ki, amelyek intenzitása különbözik a háttértől. A "Blobok" nagy intenzitású fehér képek voltak, a háttér pedig fekete. Ha engedélyeznénk egy kicsit több időt, akkor az blobítás funkció helyett egy másik Blob képkonverziót használtunk volna, hogy pontosabbak és jobban illeszkedjenek a képeinkhez, de több időre volt szükségünk a funkció kutatásához és megvalósításához.
5. lépés: A sejtek számlálása és a sejtek összefolyásának kiszámítása

A kód ezen lépésében a képen lévő cellák számának meghatározását tűztük ki célul. Elsősorban a regionprops függvényt használtuk a foltok területeinek kiszámításához, és ha a területet a kívánt határon belülre számítottuk, azt egy részterületen ábrázoltuk. A határokat úgy határozták meg, hogy eltávolítsák a kis zajos képpontokat vagy a nagy intenzitást, amelyek nem cellák. A cellaszámláló ezután megszámlálja az ezután ábrázolt középpontokat, és hozzáadja őket a for ciklus számlálójához. Miután meghatároztuk a sejtek területeit, ki tudtuk számítani az összefolyást. Ennek a lépésnek a legfontosabb jelentősége a kódban az volt, hogy megtaláljuk a cellák összefolyását, és ez kulcsfontosságú volt a kód végső célunk szempontjából. Ezt úgy végeztük, hogy úgy számoltuk, hogy összegeztük az egyes blobok pixeleit (sum (allAreas)), majd elosztottuk a kép teljes pixelértékével (numel (img)). Ez az arány megadja számunkra az összefolyást, és ha azt 80% -nál nagyobbnak határozták meg, akkor itt az ideje, hogy a kutató átadja a sejteket. Arra törekedtünk, hogy minél pontosabbak és pontosabbak legyünk, de a korlátozott idő alatt így pontatlanságok fordultak elő. Ha az idő engedi, akkor megvizsgáltuk volna a foltok számolásának pontosabb módszereit, például több szűrési technikát és/vagy Hough -transzformációt, mivel még nem végeztek elegendő kutatást a szűrési technika kipróbálására.
6. lépés: A sejtek gömbölyűsége
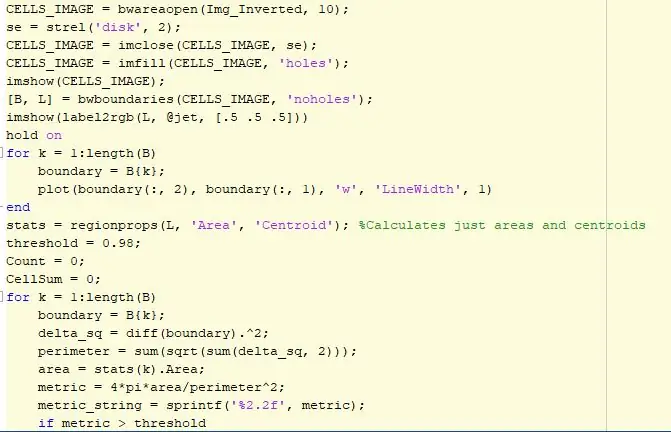

Mielőtt meg tudnánk mérni a folt gömbölyűségét a képen, át kell alakítanunk RGB -ről szürkeárnyalatosra, binárisítani, invertálni és szűrni a képet. Egy szűrési technika a bwareaopen funkciót használja, amely szűri az érdeklődésre számot tartó képet, és eltávolít minden olyan adatot vagy képpontot, amely túl kicsi, és nem reprezentálja a cella méretét. A strukturáló elem egy lemez alakú és 2 -es szomszédságú, és a háttérben vagy a cellákban lévő hiányosságok kitöltésére szolgál. Ezután a bwboundaries függvényt használjuk, amely nyomon követi a blobokat, és eltárolja egy mátrixba. Ezután különböző színekkel címkézzük meg a képet, hogy tisztább legyen a kép. Ezután egy for for ciklus segítségével, amely a képen talált objektumok és lyukak számának megfelelően fut, határt szab a tömbnek megfelelő blobok körül. Miután ez a ciklus befejeződött, egy másik ciklus kezdődik, ismét a képen található objektumok és lyukak számának megfelelően. Ezúttal a regionprops függvényt használjuk, amely bizonyos tulajdonságokat, például területet gyűjt a tömbből, és tárolja az objektumok és lyukak számát tartalmazó információkat. Ezen információk felhasználásával a középpont alakzat segítségével kiszámítjuk ezen objektumok területét és kerületét. Egy küszöbértéket állítunk össze az eredmények összehasonlítására, miután kiszámítottuk egy kör alakú objektum metrikus egységét, és ezt a mérést szövegként ábrázoltuk a számított középpontok mellett. A végső kód a képen látható különböző cellák kerekítését jeleníti meg, és az egyik értékhez közeli érték kerekebb lesz, mint mások. Az 1 érték azt jelenti, hogy a cella tökéletesen kerek és jó állapotban van ahhoz, hogy áthaladjon.
Ajánlott:
Gimbal stabilizátor projekt: 9 lépés (képekkel)

Gimbal Stabilizer Project: Hogyan készítsünk egy kardántengelyt? Ismerje meg, hogyan készítsen kéttengelyes kardántengelyt az akciókamerájához. A mai kultúrában mindannyian szeretjük a videókat rögzíteni és megörökíteni a pillanatokat, különösen akkor, ha Ön olyan tartalomkészítő, mint én, biztosan szembesült azzal a problémával, hogy ilyen remegő videó
Automatizált EKG-BME 305 Végső projekt Extra jóváírás: 7 lépés

Automatizált EKG-BME 305 A végső projekt extra jóváírása: Egy elektrokardiogram (EKG vagy EKG) a dobogó szív által kibocsátott elektromos jelek mérésére szolgál, és nagy szerepet játszik a szív- és érrendszeri betegségek diagnosztizálásában és prognózisában. Az EKG -n szerzett információk egy része tartalmazza a ritmust
(IOT projekt) Időjárási adatok lekérése az ESP8266 és az Openweather API használatával: 5 lépés

(IOT Project) Időjárási adatok lekérése az ESP8266 és az Openweather API használatával: Ebben az utasításban egy egyszerű IOT projektet fogunk felépíteni, amelyben lekérjük városunk időjárási adatait az openweather.com/api webhelyről, és megjelenítjük azokat a Feldolgozó szoftver segítségével
BME 305 EEG: 4 lépés

BME 305 EEG: Az elektroencefalogram (EEG) egy olyan eszköz, amelyet egy alany elektromos agyi aktivitásának mérésére használnak. Ezek a vizsgálatok nagyon hasznosak lehetnek a különböző agyi rendellenességek diagnosztizálásában. Amikor EEG -t készítünk, különböző paraméterekre van szükség
Homokozó projekt: BAC számítás és értelmezés: 6 lépés

Sandbox Project: BAC Calculation and Interpretation: Írta: Haarika Gogineni, Hana Schlosser és Benedict Uiseco A kiszámított BAC kiadása után megállapítjuk a
